Hochaufgelöste Einblicke in den Transport von Glutamat durch Computer-Simulationen und Röntgenkristallographie
Jülich, 19. November 2020 – Jülicher Forscher haben mithilfe von Simulationen auf dem Supercomputer JURECA und Röntgenkristallographie aufgeklärt, wie Natriumionen den Transport von Glutamat im Gehirn antreiben. Zuständig dafür sind die sogenannten Excitatory Amino Acid Transporters, kurz EAATs, welche den Botenstoff Glutamat aus dem synaptischen Spalt zwischen Nervenzellen entfernen. Der Beitrag wurde in der aktuellen Ausgabe des renommierten Fachmagazins "Science Advances" veröffentlicht.
Glutamat ermöglicht es, dass aktivierende Signale von einer Nervenzelle auf eine benachbarte übertragen werden. Damit die Signalübertragung genau mit Ende des aktivierenden Signals auch wieder aufhört, muss Glutamat nach seiner Freisetzung schnell aus der Synapse abtransportiert werden. Das ist die Aufgabe von spezialisierten Proteinen in der Zellmembran, den EAAT Glutamat-Transportern.
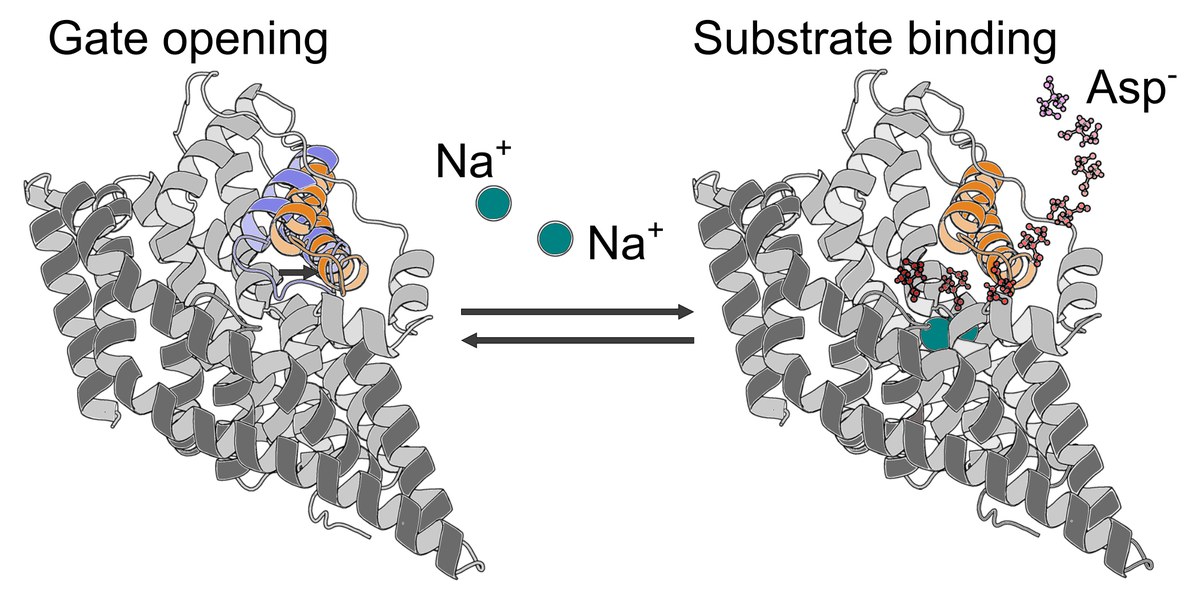
EAATs sind sekundär-aktive Transporter und nutzen das Konzentrationsgefälle von Natriumionen, um die Glutamat-Aufnahme in die Zellen anzutreiben. Die Transporter binden dazu den Neurotransmitter Glutamat an der Außenseite der Zelle zusammen mit insgesamt drei Natriumionen und verfrachten alles zusammen in das Zellinnere. Das natürliche Gefälle der Natrium-Konzentration, die außerhalb der Zelle deutlich höher ist als im Inneren, wirkt dabei als treibende Kraft.
Bislang war unklar, wie EAATs das Glutamat zusammen mit Natriumionen binden und wie die Ionen diesen Prozess antreiben. Die Jülicher Forscher haben diese Frage nun beantwortet: Mithilfe von Röntgenkristallographie gelang es, mit bislang unerreichter Genauigkeit eine hochaufgelöste Struktur eines natriumgebundenen Transporters unmittelbar vor der Bindung des Glutamats aufzunehmen. Simulationen auf dem Jülicher Supercomputer JURECA und weitere Experimente konnten zeigen, wie die Bindung von zwei Natriumionen die nachfolgende Bindung von Glutamat und eines dritten Natriumions ermöglicht.
Die Ergebnisse liefern wichtige Einblicke in molekulare Prozesse der Informationsverarbeitung im Gehirn und könnten hilfreich sein, um neue Behandlungsverfahren für ischämische Hirnerkrankungen wie den Schlaganfall zu entwickeln, bei denen erhöhte Glutamat-Konzentrationen auftreten.
Originalpublikation:
Na+-dependent gate dynamics and electrostatic attraction ensure substrate coupling in glutamate transporters, Alleva, Kovalev et al., Sci. Adv. 2020; 6: eaba9854; 18. November 2020
https://doi.org/10.1126/sciadv.aba9854
Ansprechpartner:
Jun.-Prof. Jan-Philipp Machtens
Institute of Biological Information Processing, Molekular- und Zellphysiologie (IBI-1)
Tel.: 02461 61-4043
E-Mail: j.machtens@fz-juelich.de
Prof. Christoph Fahlke
Institute of Biological Information Processing, Molekular- und Zellphysiologie (IBI-1)
Tel.: 02461 61-3016
E-Mail: c.fahlke@fz-juelich.de
Pressekontakt:
Erhard Zeiss, Pressereferent
Tel.: 02461 61-1841
E-Mail: e.zeiss@fz-juelich.de
